研究内容
研究の背景
細胞内の異常タンパク質の蓄積は、様々な細胞機能障害を引き起こすため、タンパク質恒常性機構により認識され排除されることが、正常な細胞機能の維持に必要である。翻訳の正確性を向上させ異常タンパク質の産生を未然に防ぐことは、アルツハイマー病やパーキンソン病をはじめとする一連の神経変性疾患抑制の新規かつ有効なアプローチであると考えられる。我々は、異常タンパク質蓄積により引き起こされる疾患の理解と創薬の分子基盤を明らかにすることを目的に、異常蛋白質の産生を抑制する品質管理機構の分子機構を解析している。我々は、異常な翻訳を監視し異常タンパク質を合成途中で分解除去する翻訳品質管理機構RQC (Ribosome-associated quality control)を初めて発見した。近年その分子機構と生理機能の解明が急速に進み、タンパク質恒常性維持のごく初期段階として極めて重要な役割を果たすことが明確になってきている。翻訳の正確性を上昇させるようにリボソーム機能を亢進させ、かつ翻訳品質管理機構の活性を上昇することができれば、異常タンパク質の産生を未然に防ぎ健康寿命を延長させることが可能と考えられる。
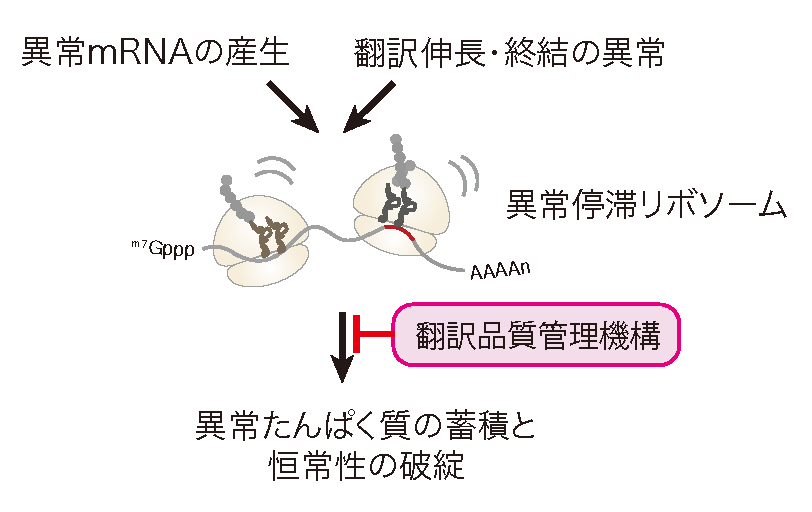
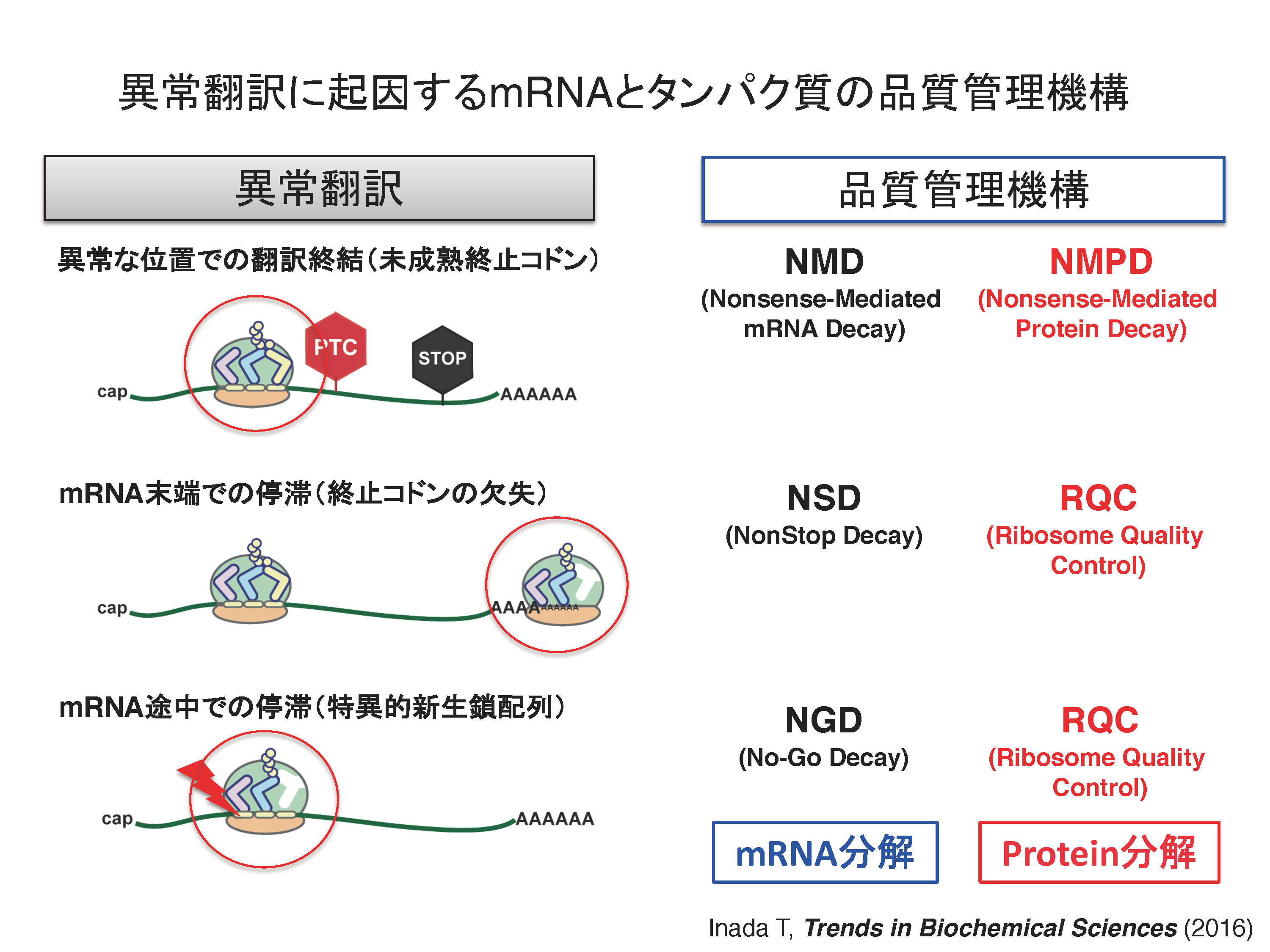
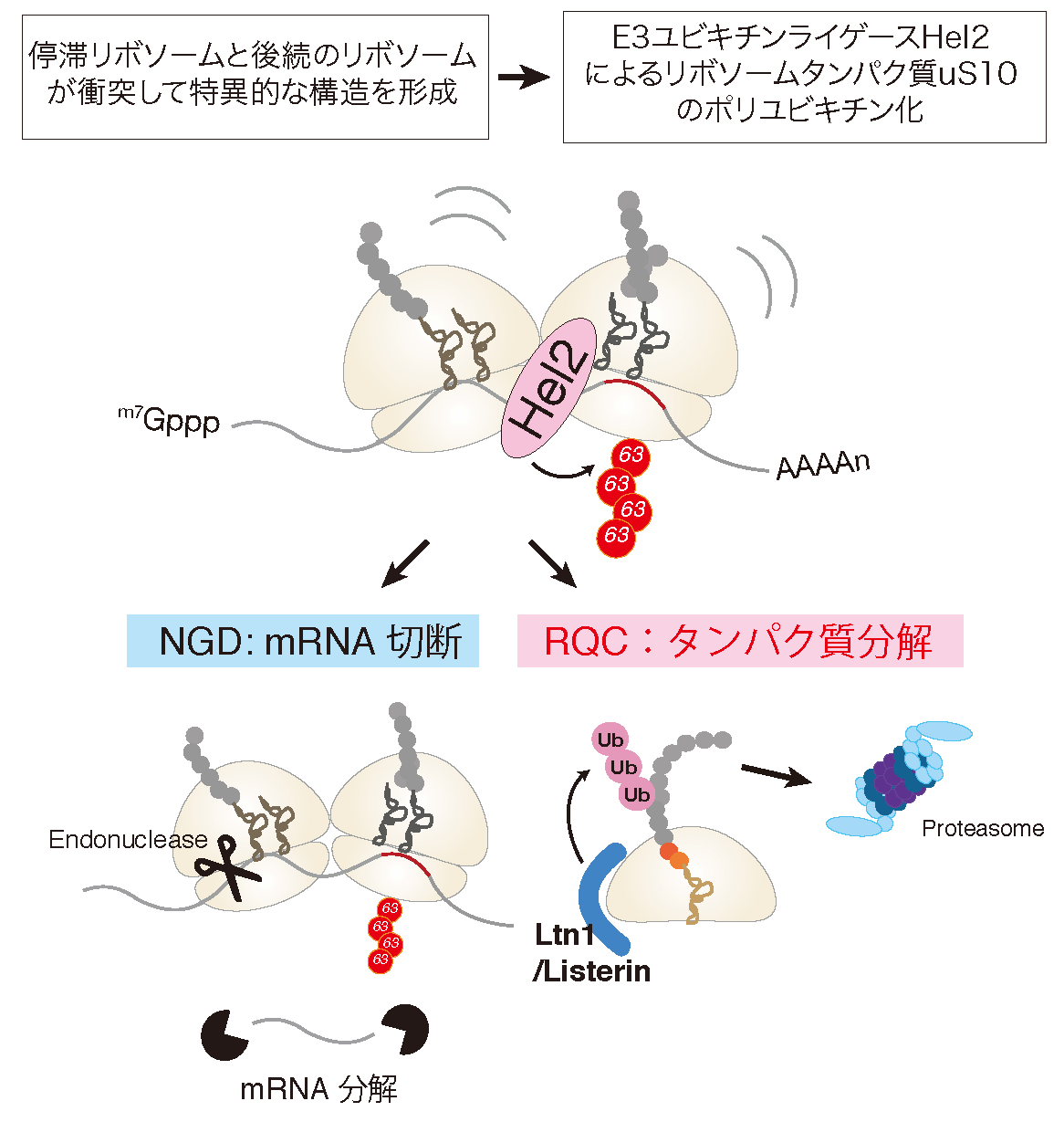
翻訳異常に対する品質管理RQCとNGDの分子機構
正確な遺伝子発現は生命現象の根幹であり、異常タンパク質の産生はタンパク質恒常性の破綻をもたらす根本原因と考えられる。異常タンパク質は、翻訳後に様々な要因で立体構造等の変化により生じるが、異常な mRNAや不正確な翻訳反応によっても生じることが明らかになってきた。異常な翻訳を認識し排除する品質管理機構によって、正確な翻訳反応が保証されると考えられてきたが、その実体は不明であった。我々は、異常翻訳により新生鎖のユビキチン化とプロテアソームによる迅速な分解が起こることを世界に先駆けて報告した(EMBO J.,2005;Genes Dev.,2007;JBC,2009)。我々の翻訳の正確性を保証する品質管理機構であるRQC(Ribosome-associated Quality Control)の発見以降、RQCがタンパク質恒常性維持のごく初期段階として極めて重要な役割を果たすことが明確になってきている(Inada, T.Trends in Biol. Sci., 2016)。我々は、RQCの初期段階である翻訳伸長中に停滞したリボソームを認識し解離させる分子機構を発見した(EMBO Rep., 2009, 2010;Mol. Cell, 2012;Cell Rep., 2012;Cell, 2012;JBC, 2014;JBC, 2015;Sci. Rep., 2016)。RQC の最も初期段階である翻訳伸長段階で停滞したリボソームの識別と各サブユニットへの解離反応の解明が急速に進んだ。まず、衝突リボソーム(Disome/Trisome)を構造決定し、E3ユビキチンライゲース Hel2が認識しする翻訳伸長中に停滞したリボソームの特異的な構造を明らかにした。kE3ユビキチンライゲースZNF598によるuS10のユビキチン化を再構成した(EMBO J., 2019; NSMB, 2020)。次に、ユビキチン化リボソーム特異的にサビユニットに解離するRQT複合体を同定し試験管内で反応を再構成した (NSMB, 2020; Sci. Rep. 2020)。また、60S上のpeptidyl-tRNAを切断するANKZF1と60Sリボソームの構造を解明した(Nature, 2019)。NGD(No-Go Decay)は翻訳停滞で誘起されるmRNAの分子内切断反応であり、RQCと共役して起こる品質管理機構である。研究代表者は、RQCでの停滞したリボソームの乖離反応に共役するmRNA切断とRQCとは独立したmRNA切断の2つの経路が存在することを発見した(EMBO J., 2019)。
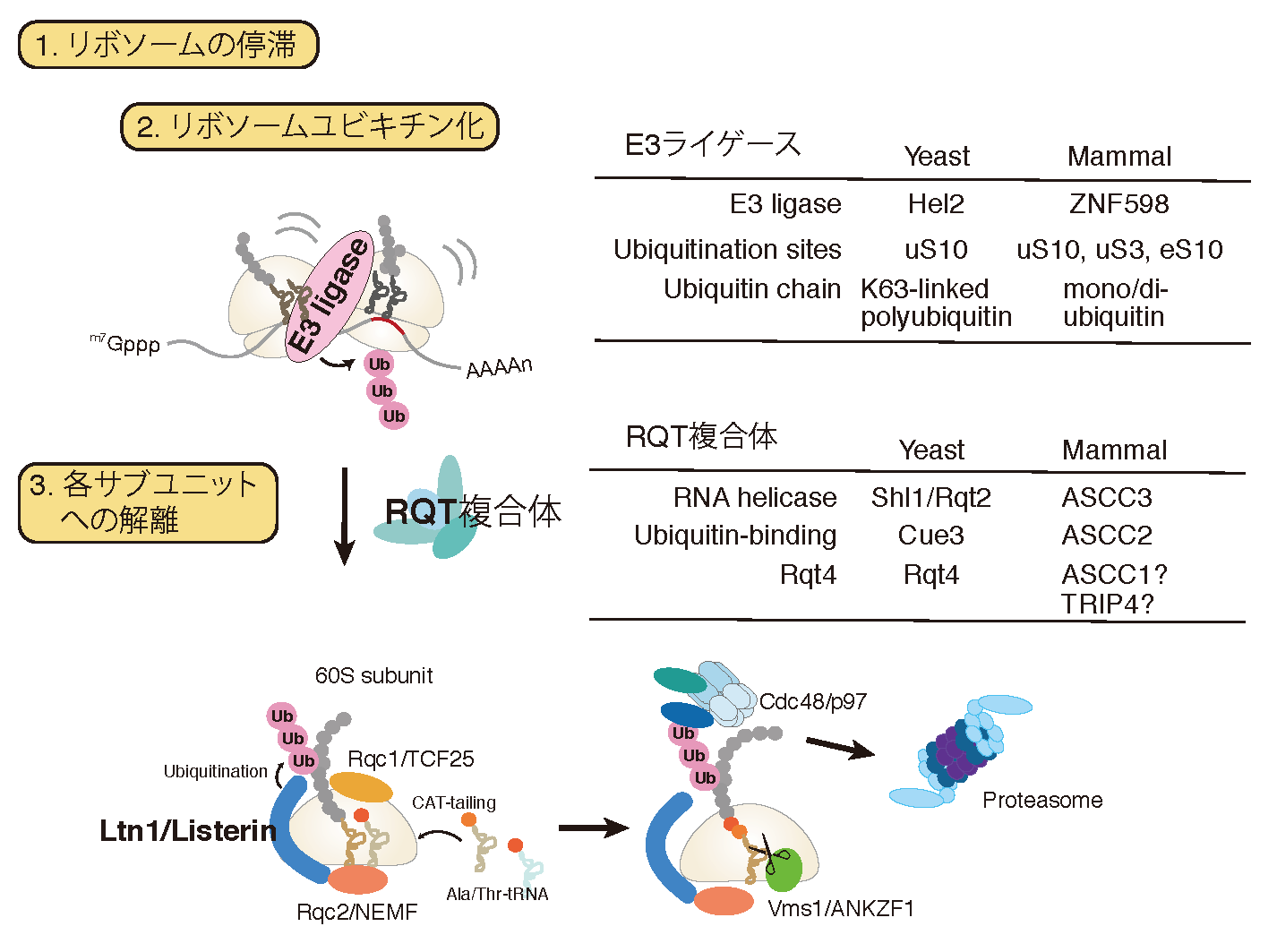
機能欠損リボソーム対する品質管理NRDの分子機構
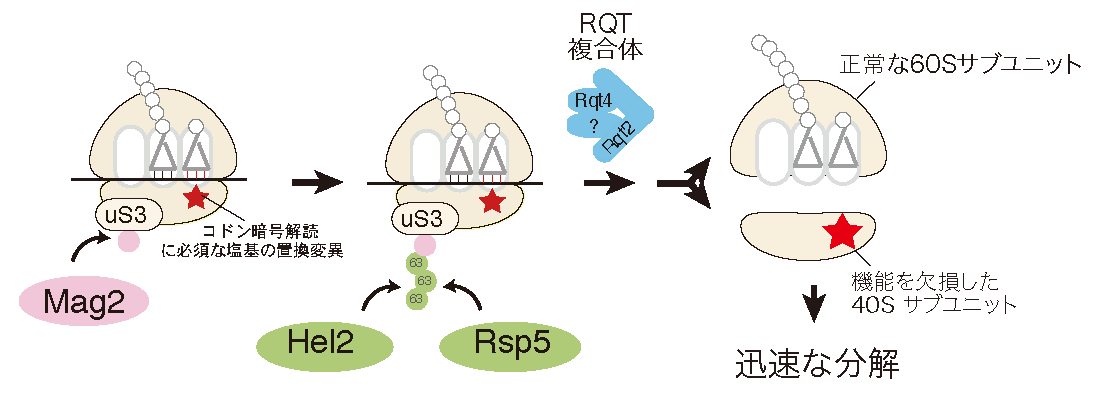
リボソームは、正確なコドン認識と高効率なペプチド結合を担うタンパク合成の中心装置であるだけでなく、様々な因子と相互作用し遺伝子発現に必須な機能を果たす。一方で、リボソーム合成は細胞のエネルギーの多くを消費し数百の因子が関与する、細胞内の主要な反応である。その合成途上で生じた異常リボソームは様々な発現異常の原因となるため、細胞は機能欠損リボソームを認識し排除する品質管理機構Nonfunctional Ribosomal RNA Decay(NRD)を保持している。我々は最近18S rRNA内の正確なコドン認識に必須な、全ての生物種で保存されている 塩基の置換変異によって機能を欠損したリボソームの品質管理を解析し、リボソームタンパク質uS3のK212残基のユビキチン化が必須であり、関与する E3 ユビキチンライゲースの同定に成功した(Cell Rep., 2019)。ユビキチン化された停滞した80Sリボソームは、RQT2によってを各サブユニットに解離されたのち、異常な40Sのみが分解されることも明らかにした。
ストレス応答におけるリボソーム動的修飾の機能
小胞体における分泌蛋白質の合成と修飾は細胞に必須である。小胞体内での異常蛋白質の蓄積は細胞に有害であるため、UPR経路を誘導することで応答する。出芽酵母では、小胞体ストレスにより活性化された膜蛋白質Ire1が、転写因子 Hac1の前駆体 mRNAをスプライシングし、Hac1が合成されてシャペロン等の転写が誘導される。高等真核生物でのでは、 PARKによってeIF2αがリン酸化され細胞全体の翻訳開始が抑制される。リボソームユビキチン化の生理機能を解明する過程で、我々は小胞体ストレス応答における新規翻訳制御を発見した。出芽酵母における小胞体ストレス時の新規な翻訳制御機構を発見し、E3ユビキチンライゲースNot4によるリボソームタンパク質eS7のユビキチン化が必須であることを明らかにした(BBRC 2020; Sci. Rep. 2020)。
翻訳品質管理の生理機能とその異常による疾患
正確な遺伝子発現は生命現象の根幹であり、異常タンパク質の産生はタンパク質恒常性の破綻をもたらす根本原因である。翻訳の正確性を向上させ異常タンパク質の産生を未然に防ぐことは、アルツハイマー病やパーキンソン病をはじめとする一連の神経変性疾患抑制の新規かつ有効なアプローチである可能性がある。遺伝子発現の根幹であるタンパク質合成装置であるリボソーム機能の低下は、不良タンパク質の蓄積やオルガネラの損傷、シグナル伝達経路撹乱など、広範な細胞機能障害を引き起こすため、個体寿命の短縮の原因になると考えられる。しかしながら、リボソーム機能低下が細胞老化・個体機能低下を引き起こす過程はいまだ不明のままである。我々は、正確なコドン識別能を欠損したリボソームに対する品質管理機構NRD(Non-functional rRNA Decay)を解析し、機能欠損リボソームの認識・排除に必須なE3ユビキチンライゲースを発見した。リボソーム機能の異常を認識し排除するシステムの実体解明を基盤に、翻訳の正確性を向上される手法の開発を試みる。
mRNAの安定性を決定する新たな分子機構の発見
我々は、mRNA安定性決定機構としてCcr4-NOTが翻訳伸長速度を感知して分解速度を決定する分子基盤を明らかにした(Science, 2020)。
【背景】
mRNAにコードされる遺伝情報は、リボソームによってタンパク質へと変換されます。この際、リボソームはmRNA上の3つの塩基配列を1つの読み枠(コドン)として遺伝情報を解読しますが、ほとんどのアミノ酸は複数種類のコドンによってコードされています。同じアミノ酸に対応するそれぞれのコドンは同義コドンとよばれ、対応するtRNAの細胞内存在量には偏りがあります。多く使われるコドンは最適コドンと呼ばれ、同義コドンが最適かどうかがは、対応するtRNAの存在量に従って数値化され、tRNA量が高いほど最適度が高いと評価されます。従って、最適度が高いコドンが多いmRNAを翻訳するときの伸長速度は速くなり、合成されるタンパク質も多いことが知られています。翻訳の伸長速度は発現量の調節だけでなく、合成されるペプチド鎖のフォールディングやターゲティングなどとも密接に連動しているため、コドンの選択は遺伝子発現において非常に重要な役割を担っています。
近年、個々のmRNAがもつ固有の安定性は、コドンの最適度によって調節されていることが報告されました。最適度が高いコドンを持つmRNAほど安定であり、最適度が低いコドンを持つmRNAは不安定であるという一般則が確立されました。最適度が高いコドンを持つmRNAほど翻訳の伸長速度が早いため、コドンの最適度によってmRNAの半減期が決定されることになります。一方で、 コドンの最適度によって調節される翻訳の伸長速度を監視し、個々のmRNAがもつ固有の安定性を決定する機構は依然として不明なままでした。
【成果】
mRNAの転写・分解や翻訳抑制に関与するCcr4-Not複合体は、RNA結合タンパク質を介してmRNAに結合することが広く知られていますが、我々は、はじめに生化学的手法を用いて、Ccr4-Not複合体がリボソームに直接結合することを見いだしました。続いて、Ccr4-Not複合体が特異的に結合するリボソームによって翻訳されるmRNAの特徴を調べるために、選択的リボソームプロファイリングによる網羅的解析を行いました。その結果、コドンレベルの解析では、Ccr4-Not複合体とリボソームの親和性がコドンの最適度と非常に強い逆相関を示すことがわかりました。(図1)
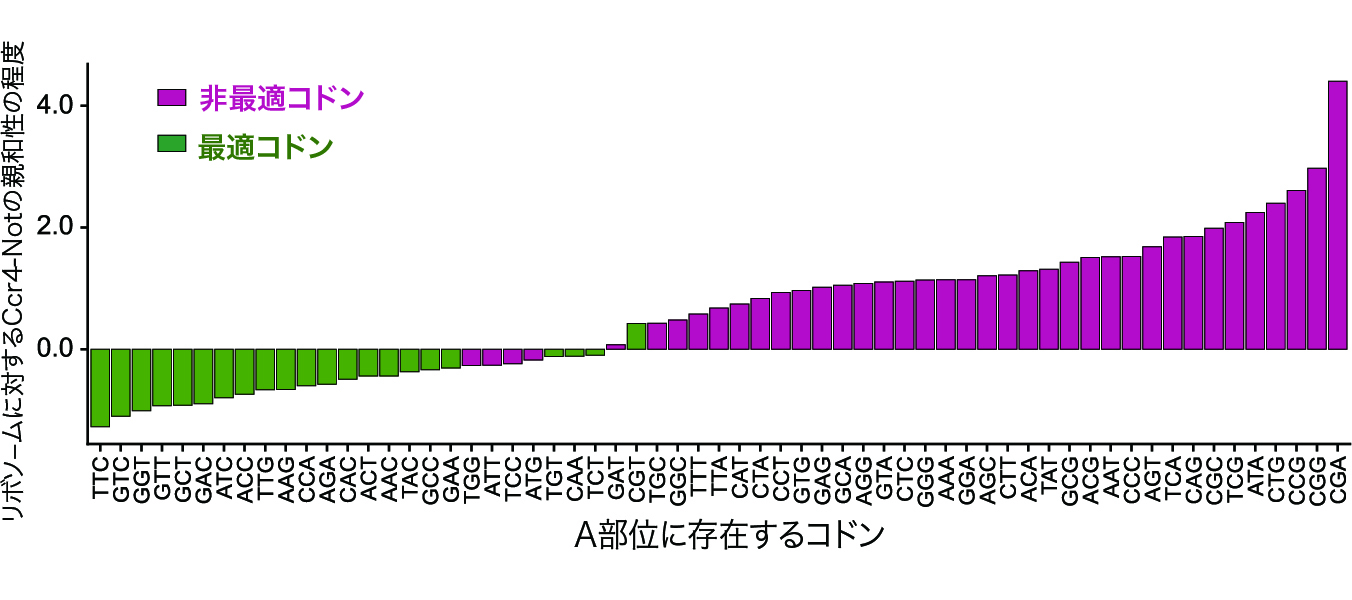
図1 Ccr4-Notとリボソームの親和性は最適コドンに強い逆相関を示す。
縦軸はリボソームに対するCcr4-Not複合体の親和性を、横軸はリボソームのA部位のコドンを示す。非最適コドン(赤)を含むリボソームはCcr4-Not複合体に高い親和性を示し、最適コドン(緑)を含むリボソームは低い親和性を示した。従って、Ccr4-Not複合体が結合するリボソームのA部位には、より最適度の低いコドンが存在することが明らかになった。
また、我々はCcr4-Not複合体の機能欠損によって、コドンの最適化によるmRNAの安定性制御が失われることも示しました。つまり、コドンの最適度が高い(最適コドンが多い)mRNAでおこる安定化や、コドンの最適度が低い(最適コドンが少ない)mRNAでおこる不安定化がみられなくなりました。以上の結果より、Ccr4-Not複合体は、コドンの最適度が低いmRNAを翻訳するリボソームに対して強い親和性をもつことで、mRNAを分解に導くことが明らかになりました。
次に、我々は、リボソームに結合したCcr4-Not複合体の構造を決定しました。クライオ電子顕微鏡を用いた単粒子解析で、Ccr4-Not複合体の構成タンパク質の1つであるNot5が、A部位にtRNAを含まないリボソームのE部位に結合することを見いだしました(図2)。翻訳過程において、コドン-アンチコドンの認識はリボソームのA部位によって行われます。非最適コドンを翻訳しているリボソームでは、対応するtRNA の存在量が少ないため、A部位にtRNAが結合するまでに長い時間が必要となります。A部位にtRNAが結合しないため、E部位からtRNAが解離します。tRNAが解離し空になったE部位にCcr4-Not複合体が結合し、効率よくmRNAを分解に導くことがわかりました。 以上の結果より、Ccr4-Not複合体は、非最適コドンを翻訳中のリボソームのE部位に効率よく結合することで、コドンの最適度を監視し、mRNAの安定性を制御することが明らかになりました(図3)。

図2 Not5とリボソームの結合様式
左図:クライオ電子顕微鏡注12を用いたCcr4-Not複合体とA部位にtRNAを含まないリボソームの単粒子解析。Ccr4-Not複合体の構成タンパク質の1つであるNot5が、リボソームのE部位に結合している。ピンクはNot5のアミノ末端領域、緑はtRNA、黄色は40Sリボソーマルサブユニット、灰色は60Sリボソーマルサブユニットを示す。
右図:リボソームのA部位に非最適コドンが存在する場合、Not5がE部位に結合することで mRNAの分解を引き起こす。
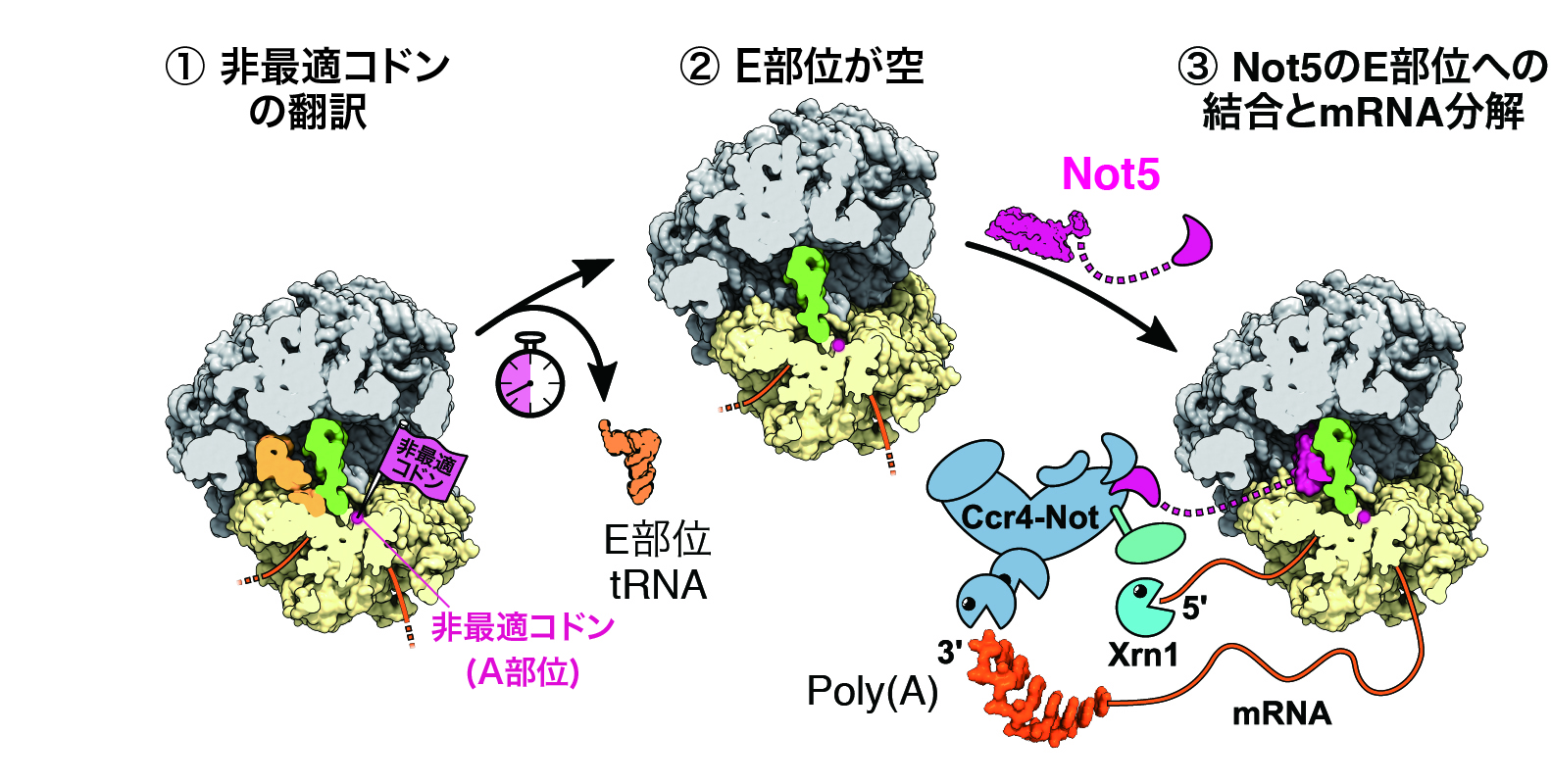
図3 コドンの最適度に依存したmRNAの分解制御機構モデル
① 非最適コドンがA部位に位置したリボソーム。tRNAの存在量が少ないため、A部位にtRNAが結合するまでの時間が長い。② A部位にtRNAが結合しないため、E部位からtRNAが解離する。③Ccr4-NOT複合体のNot5 サブユニットのN末端領域がE部位に結合する。リボソームに結合したCcr4-NOT複合体がポリAを短鎖化する。さらにキャップ構造の除去後にリボヌクレアーゼXrn1がmRNAを分解する。ピンクはNot5のアミノ末端領域、マジェンダはE部位に結合したtRNA、緑はP部位に結合したtRNA、黄色は40Sリボソーマルサブユニット、灰色は60Sリボソーマルサブユニット、オレンジは mRNAを示す。
【まとめ】
本研究により、コドンの最適化によるmRNAの分解制御機構の実態が明らかになり、長年不明であった遺伝情報における同義コドンの存在意義がより明確になりました。タンパク質合成途中の翻訳速度調節の異常は、タンパク質の機能に重大な欠陥を引き起こし、タンパク質恒常性の破綻につながります。タンパク質恒常性の破綻は、不良タンパク質の蓄積やオルガネラの損傷、シグナル伝達経路のかく乱など、広範な細胞機能の障害を引き起こすため、アルツハイマー病やパーキンソン病などの神経変性疾患や老化の原因になると考えられます。今回の研究成果は、翻訳異常によって機能欠損タンパク質が合成されることで発症する疾患の発症機構や老化のメカニズムを理解するための基盤になることが期待されます。